Alles Gute
Protein-Landkarte

Bild: ediundsepp Gestaltungsgesellschaft, München

Bild: ediundsepp Gestaltungsgesellschaft, München
Sie bestimmen die Form unserer Haare, verdauen unser Essen und kämpfen gegen Krankheitserreger: Ohne Proteine läuft im Körper nichts. Ein Forschungsteam der Technischen Universität München (TUM) identifiziert 18.097 dieser Eiweiße und damit fast alle Proteine des Menschen. In Zukunft soll so für jeden Menschen eine individuelle Therapie möglich werden – auch gegen Krankheiten wie Krebs.
In Speichel, Blut und Ohrenschmalz suchen sie nach den Alleskönnern unseres Körpers: den Proteinen. Sie durchforschen unterschiedlichste Körpergewebe und Körperflüssigkeiten, sogar zahlreiche Tumorzellen: So erstellen Wissenschaftlerinnen und Wissenschaftler um den Biochemiker Bernhard Küster 2014 die erste Karte des menschlichen Proteoms, also der Gesamtheit aller Proteine in unserem Körper. Sie identifizieren 18.097 dieser Eiweiße und damit über 90 Prozent des Proteoms. Und sie finden heraus, wo sie sich im Körper befinden und wie viele Varianten es jeweils von ihnen gibt.
Das ist wichtig für die Entwicklung einer so genannten personalisierten Medizin, die speziell auf die Patientin oder den Patienten ausgerichtet ist. Denn was in den vergangenen Jahren immer klarer wurde: Nicht nur unsere Gene, sondern auch unsere Proteine haben Einfluss auf uns und unsere Krankheiten. Das gilt besonders für Krebs. Denn Tumore entstehen aus fehlgesteuerten Körperzellen, die von Patient zu Patient sehr verschieden sind. Eine personalisierte Medizin berücksichtigt somit neben den genetischen Voraussetzungen auch das Proteom von Betroffenen. Bei den Krebszellen deuten erste Untersuchungen von Küster und seinem Forschungsteam darauf hin, dass Krebsmedikamente bei Zellen von unterschiedlichen Personen anders wirken – eben wegen ihrer unterschiedlichen Proteine.
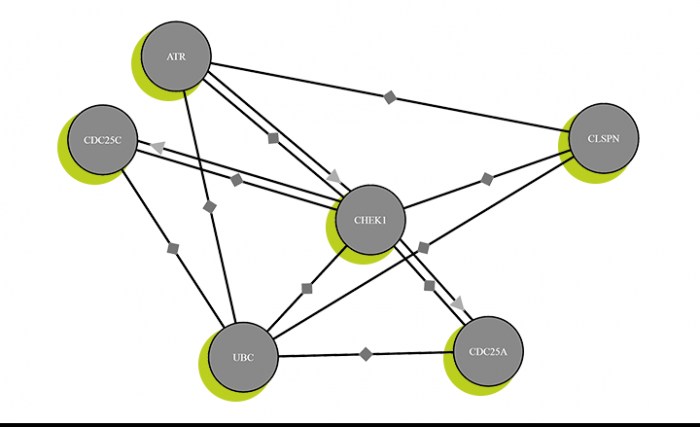
Küster und sein Team erstellen mit ihren Ergebnissen die bisher umfangreichste Protein-Datenbank „ProteomicsDB“. Mit einer neu entwickelten Software können sie selbst und Proteinfachleute weltweit ihre Daten speichern, verknüpfen und auswerten. Jeder kann die Ergebnisse der eigenen Proteinforschung hinzufügen, denn das Programm ist frei im Netz verfügbar. So füllen sie alle gemeinsam nach und nach die letzten weißen Flecken auf der Proteinlandkarte des Menschen.
Proteine sind wahre Verwandlungskünstler, das macht ihre Analyse sehr schwierig. Abhängig davon, wo und unter welchen Bedingungen sie im Körper zu finden sind, schlüpfen sie in unterschiedliche Rollen. Ihre chemischen Eigenschaften und ihr Aussehen verändern sich dann – und es wird noch schwerer, sie zu unterscheiden. Somit gibt es für jedes der fast 20.000 Proteine, die Küster und sein Team gefunden haben, noch unzählige Varianten. Die Aufgabe der Forscherinnen und Forscher wird es in Zukunft sein, auch all diese Varianten zu finden.

Proteine sind sehr klein. Deshalb sind aufwändige technische Verfahren nötig, um sie zu analysieren. Eines davon ist die Massenspektrometrie. Dabei wird zuerst die Molekülkette eines Proteins in kurze Fragmente zerlegt, um diese dann nach Gewicht und elektrischer Ladung zu sortieren. Wie bei einem Puzzle lassen sich anschließend die Daten auswerten, die über die Einzelstücke gewonnen werden, und am Computer wieder virtuell zusammenfügen. Im Vergleich mit bereits bekannten Datensätzen können Forscherinnen und Forscher dann sehr genau bestimmen, um welches Protein es sich handelt. Entscheidend dafür sind aber große Datenbanken, die alle bisher bekannten Proteine verzeichnen – so wie die Datenbank von Bernhard Küster.
„Wenn wir zum Beispiel das Proteinprofil eines Tumors kennen, könnten wir in Zukunft Medikamente viel zielgerichteter verabreichen.“

Bernhard Küster, Professor für Proteomik und Bioanalytik an der Technischen Universität München
Bild: Filser